偶氮染料废水的脱色方法主要包括物化法和生物法〔1〕。常见的物化法有萃取法、吸附法、混凝法、膜过滤法、化学氧化法、电化学氧化法等,这些方法大多存在成本高、能耗大、易产生大量污泥和有毒副产物等缺点,易造成二次污染〔5〕。生物法因成本低、运行稳定且环境友好,成为偶氮染料废水脱色的主要处置方式〔6〕。常见的脱色生物有大型水生植物〔7〕、藻类〔8〕、真菌〔9〕、酵母〔10〕和细菌〔11〕等。其中,细菌因生长周期短、生长速率高、环境适应性强,成为偶氮染料生物处理的研究热点之一。但因细菌对生存条件及营养物质要求不尽相同,温度、盐度、pH等环境因素会严重影响微生物的脱色能力,极大限制了染料脱色功能菌的工业化应用。获得高效耐受性脱色菌并强化生物系统的脱色效果,是克服生物法处理限制、解决偶氮染料废水脱色的有效途径〔5〕。研究表明,混合菌群比单一脱色菌对染料具有更好的降解作用,菌群通过协同代谢作用,可使染料降解种类更多样、产物更简单、脱色更彻底,耐受性能更广泛〔12-13〕。
垃圾渗滤液成分复杂、水质水量波动大、盐度和重金属含量高〔14〕,对处理系统中微生物的耐冲击能力要求高。本研究拟从运行良好的垃圾渗滤液生化处理系统的活性污泥中富集筛选高效脱色菌并进行复配,考察复配组合的脱色效能,并对复配菌群的普适性和耐冲击能力进行评估,以期为高盐高碱度染料废水的深度脱色提供微生物种质资源。
1 实验部分
1.1 实验材料
(1) 污泥来源。南京市某垃圾焚烧厂垃圾渗滤液生化处理系统的活性污泥。
(2) 染料。中性红、甲基橙、刚果红、亚甲基蓝、孔雀石绿、活性黑5、活性艳红X-3B,均为分析纯。
(3) 培养基。
富集培养基:蛋白胨2.0 g/L,Na2HPO4 1.35 g/L,KH2PO4 1.8 g/L,葡萄糖5.0 g/L,MgSO4·7H2O 0.2 g/L,活性艳红X-3B 0.025~0.1 g/L,pH 6.5~7.5;
分离纯化培养基:蛋白胨2.0 g/L,Na2HPO4 1.35 g/L,KH2PO4 1.8 g/L,葡萄糖5.0 g/L,MgSO4·7H2O 0.2 g/L,活性艳红X-3B 0.1 g/L,琼脂20 g/L,pH 6.5~ 7.5,121 ℃灭菌后备用;
LB培养基:酵母粉5.0 g/L,氯化钠10 g/L,蛋白胨10 g/L,pH 7.0~7.5,121 ℃灭菌后备用;
活性艳红X-3B脱色培养基:蛋白胨2.0 g/L,Na2HPO4 1.35 g/L,KH2PO4 1.8 g/L,葡萄糖5.0 g/L,MgSO4·7H2O 0.2 g/L,活性艳红X-3B 0.1 g/L,pH 6.5~ 7.5,121 ℃灭菌后备用。
(4) 染料配水。蛋白胨2.0 g/L,Na2HPO4 1.35 g/L,KH2PO4 1.8 g/L,葡萄糖5.0 g/L,MgSO4·7H2O 0.2 g/L,染料0.1 g/L,pH 6.5~7.5。
1.2 实验方法
1.2.1 菌株的驯化富集、分离纯化及脱色性能评估
取垃圾渗滤液处理系统的好氧段活性污泥和厌氧段活性污泥各10 g分别接种至100 mL富集培养基(活性艳红X-3B质量浓度为25 mg/L)中,于生化培养箱中35 ℃静置培养,至培养基呈现无色后,得到一级培养液,吸取一级培养液10 mL接种至新的富集培养基中(活性艳红X-3B质量浓度为50 mg/L),同上操作,获得二级培养液,至活性艳红X-3B质量浓度提高至100 mg/L,获得三级培养液。取三级培养液按5%接种量在活性艳红X-3B质量浓度100 mg/L的富集培养基中连续培养至脱色,驯化3次,完成富集过程。
采用梯度稀释法将富集的菌液稀释10~106倍,取稀释液均匀涂布于筛选培养基上,35 ℃倒置培养,挑取生长较快的菌落多次划线,分离纯化得到单菌。
将筛选获得的单一脱色菌株接种于LB培养基中,35 ℃下振荡培养24 h,得到单一菌株的扩大培养菌液。按5%接种量分别接种于灭菌后的活性艳红X-3B脱色培养基中,35 ℃下厌氧静置培养,定期测定单一菌株在538 nm处的吸光度,依照式(1)计算脱色率〔15〕。

式中:Aλmax——染料最大吸收峰脱色前的吸光度;
Bλmax——染料最大吸收峰脱色后的吸光度。
1.2.2 菌株的鉴定
将筛选获得的高效脱色菌于LB固体培养基上生长,观察记录菌落形态、大小,并通过革兰氏染色法〔16〕进行初步鉴定。
1.2.3 复配菌株脱色实验
分别将筛选获得的高效脱色菌株于LB培养基中35 ℃振荡培养24 h得到菌株母液,按5%总接种量,采用等体积进行复配组合,并将不同复配混合液分别接种至活性艳红X-3B脱色培养基中,于35 ℃下培养,测定反应液在538 nm处的吸光度,对比分析不同复配组合对活性艳红X-3B的脱色速率。
1.2.4 复配菌株适应性研究
将最佳复配组合混合液按接种量5%分别接种于初始pH为7、8、9、10的活性艳红X-3B脱色培养基中,以不添加复配菌株为对照组,静置培养48 h,记录处理前后吸光度及pH变化,并计算脱色率。
以NaCl调整活性艳红X-3B脱色培养基盐度分别为0%、3%、5%、10%、15%,将最佳复配组合混合液按接种量5%分别接种于不同盐度的活性艳红X-3B脱色培养基中,35 ℃静置培养至吸光度值稳定,记录处理前及稳定后的吸光度值,计算脱色率。
1.2.5 复配菌株广谱性研究
将最佳复配组合混合液按接种量5%分别接种于不同染料配水中,35 ℃静置培养72 h,测定复配菌株对不同染料的脱色率,不同染料的分子结构及最大吸收峰见表 1。
表1 染料的性质尧种类尧最大吸收峰尧分子质量及分子结构
| 染料名称 | 分类 | 最大吸收峰/nm | 相对分子质量 | 分子结构 |
| 中性红 | 碱性醌亚胺染料 | 479.5 | 288.78 | 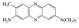 |
| 甲基橙 | 酸性偶氮染料 | 464 | 327.33 |  |
| 刚果红 | 直接偶氮染料 | 486 | 696.68 | 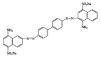 |
| 亚甲基蓝 | 碱性吩噻嗪染料 | 664 | 373.12 |  |
| 孔雀石绿 | 阴离子三苯甲烷染料 | 617 | 364.92 | 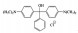 |
| 活性艳红X-3B | 活性偶氮染料 | 538 | 615.33 |  |
| 活性黑5 | 活性偶氮染料 | 598 | 991.82 |  |
2 结果与讨论
2.1 菌株的驯化富集、分离纯化与脱色性能评估
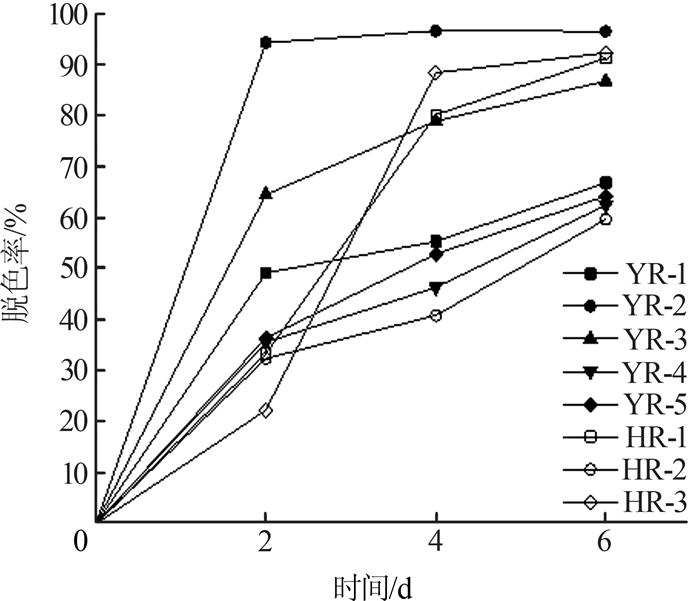
经过富集培养和初筛,从2种活性污泥中获得8株活性艳红X-3B脱色菌株,分别命名为YR-1、YR-2、YR-3、YR-4、YR-5、HR-1、HR-2、HR-3。其中,Y系列菌株来源于垃圾渗滤液厌氧段活性污泥,H系列菌株来源于垃圾渗滤液好氧段活性污泥。好氧条件下,氧气是微生物还原酶主要的电子受体,阻碍了偶氮键的裂解,此时细菌对染料的脱色具有高度的结构特异性和较长的时间适应性〔19〕;厌氧条件下,细菌在偶氮还原酶的作用下使偶氮键(—N=N—)裂解,实现脱色,此时微生物脱色具有非特异性,同时比好氧脱色具有更高的脱色效率〔11〕。因此,将筛选获得的菌株均置于厌氧条件下进行脱色处理,得到单一菌株对活性艳红X-3B的脱色效果,结果见图 1。
图1
由图 1可知,Y系列脱色菌株培养2 d后,YR-2、YR-3的脱色效果较好,脱色率均高于60%,其中菌株YR-2的脱色率高达94.34%,培养6 d,二者的脱色率分别为96.59%和86.18%,此时YR-1、YR-4及YR-5的脱色率分别为66.89%、62.47%和64.28%;H系列菌株前期脱色效果不明显,但培养至4 d时,菌株HR-1和HR-3脱色率分别由33.36%、22.34%提升至80.22%和88.44%,培养6 d,二者的脱色率分别提高至91.31%和92.32%。J. K. Thakur等〔20〕利用芽孢杆菌Azo 1对活性红HE7B进行脱色研究,结果表明,当温度为30 ℃时,该菌株对25 mg/L和50 mg/L的染料脱色率在第8 d高达100%,但随着染料浓度的增加,脱色效率显著降低,当染料质量浓度为100 mg/L时,脱色率最高仅为89%。由此可见,与已有研究相比,本研究从垃圾渗滤液生化处理系统活性污泥中筛选获得的单一菌株(YR-2、YR-3、HR-1、HR-3)对高浓度偶氮染料(100 mg/L)具有更高适应性和更好脱色效果,其中YR-2脱色效率最高,HR-3、HR-1、YR-3次之。
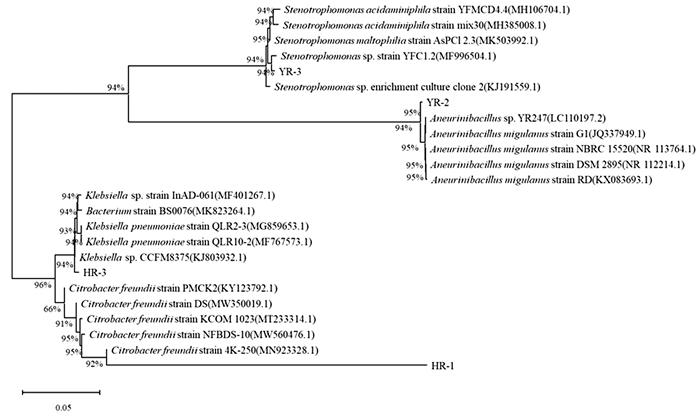
2.2 菌株的鉴定
将菌株YR-2、YR-3、HR-1、HR-3于LB培养基上划线培养,对菌落的形态和生理生化特性进行了鉴定,结果如表 2所示。将YR-2、YR-3、HR-1、HR-3的16S rDNA基因进行扩增测序,得到4种菌株的16S rDNA基因序列,在NCBI核酸数据库中进行同源性比对,选择基因相似度99%以上的序列,构建系统发育树,结果如图 2所示,菌株YR-2、YR-3、HR-1、HR-3分别为解硫胺素芽孢杆菌(Aneurini-bacillus sp.)、微嗜酸寡养单胞菌(Stenotrophomonas acidaminiphila)、弗氏柠檬酸杆菌(Citrobacter freun-dii)和克雷伯氏菌(Klebsiella sp.)。
表2 菌株YR-2尧YR-3尧HR-1尧HR-3的形态及生理生化特性
| 菌株 | 菌落特征 | 革兰氏染色 | 细胞特征 |
| YR-2 | 乳白色、不透明、边缘不整齐、表面干燥 | 阳性 | 杆菌 |
| YR-3 | 黄白色、不透明,边缘整齐、表面湿润 | 阴性 | 杆菌 |
| HR-1 | 灰白色、不透明、边缘不整齐,表面有黏性 | 阴性 | 杆菌 |
| HR-3 | 灰白色、不透明、边缘整齐,表面有黏性 | 阴性 | 杆菌 |
图2
芽孢杆菌是严格好氧或兼性厌氧且有荚膜的杆菌,不利条件下能产生具有特殊抵抗力的芽孢,使得其对恶劣环境具有一定耐受能力。S. Lalnunhlimi等〔21〕对从含盐土壤中分离获得的4株芽孢杆菌进行偶氮染料脱色研究,结果表明4株菌株不仅具有一定耐盐性,还具有较强的耐碱性,在8~10的pH范围内对直接蓝151和直接红31混合染料的脱色率达78%~85%。寡养单胞菌是一类好氧/兼性厌氧革兰氏阴性菌〔22〕。目前寡养单胞菌在染料脱色方面的研究相对较少,其脱色机理尚不明确,但通过对其解毒机理与效能的研究,发现其解毒的关键在于该菌含有漆酶基因〔23〕,该基因可能同样有助于染料的脱色降解。弗氏柠檬酸杆菌是一种需氧或兼性厌氧的革兰氏阴性菌,广泛分布于自然界中。芦银香等〔24〕从市政污泥中分离获得一株弗氏柠檬酸杆菌,对分散蓝2BLN的最高脱色率可达86.10%,但在碱性环境(pH为8~11)中该菌的生长受到严重抑制。克雷伯氏菌是一种被广泛报道的脱色菌,已从许多不同的生境(如活性污泥、工业废水等)中被分离出来〔25〕。S. Dixit等〔11〕从印染厂活性污泥中分离出一株肺炎克雷伯氏菌株,该菌能有效降解磺化偶氮染料,通过对脱色液的紫外可见光谱图分析,证实了该菌能进一步降解中间代谢产物4-氨基苯磺酸,从而降低偶氮染料脱色过程产生的中间产物的毒性。
2.3 复配菌群的脱色效能分析
已有研究显示,复配菌群可通过共代谢作用显著提高脱色效率,与单一菌株相比,脱色速率更快、适应性更强。杨财容等〔26〕从活性污泥中分离得到6株活性黑脱色菌,经复配实验组合成不同复合菌群,结果表明Y1+Y5组合的复合菌群脱色率最高,72 h脱色率为95.68%,而单一菌株最高脱色率仅为83.23%。曾丽璇等〔27〕在印染废水处理水解池中分离得到10株菌株,并对这10株菌株进行单株及混合培养脱色,结果表明,分别属于10个菌属的10株菌构成的混合菌群的脱色率显著高于单菌,且对条件适应性更强、脱色时间更短。Yan Chen等〔28〕发现包含4种厌氧菌与5种好氧菌的多菌种组合体系,不仅可以在厌氧条件下断裂偶氮键,还可以将其中间代谢产物进一步降解。通过对反应过程中酶系的进一步探究,发现这9种菌可进行协同作用产生更多的酶(漆酶、木质素过氧化物酶及锰过氧化物酶等)实现细菌之间的共代谢,有助于染料脱色。
为进一步提高生物脱色的效能,将筛选获得的4种脱色菌株按等体积进行复配,不同组合及其对活性艳红X-3B的脱色效率见表 3。
表3 不同菌株复配组合(体积比)及脱色率
| 菌株组合 | 脱色率/% | |
| 24 h | 48 h | |
| YR-2∶YR-3(1∶1) | 64.10 | 93.45 |
| YR-2∶HR-1(1∶1) | 48.59 | 62.94 |
| YR-2∶HR-3(1∶1) | 54.23 | 95.27 |
| YR-3∶HR-1(1∶1) | 45.62 | 66.82 |
| YR-3∶HR-3(1∶1) | 63.82 | 96.95 |
| HR-1∶HR-3(1∶1) | 58.65 | 97.68 |
| YR-2∶YR-3∶HR-3(1∶1∶1) | 67.08 | 97.18 |
| YR-2∶YR-3∶HR-1(1∶1∶1) | 49.18 | 87.23 |
| YR-2∶HR-1∶HR-3(1∶1∶1) | 54.56 | 92.21 |
| YR-3:HR-1:HR-3(1∶1∶1) | 59.37 | 97.03 |
| YR-2∶YR-3∶HR-1∶HR-3(1∶1∶1∶1) | 48.67 | 96.02 |
由表 3可以看出,相比单一菌株,复配菌群的脱色速率均有显著提高,其中含菌株HR-3的复配组合在48 h内脱色率均能达到92%以上。HR-1+HR-3、YR-2+YR-3+HR-3、YR-3+HR-1+HR-3三个处理组脱色效果显著,脱色率均高达97%以上。其中,YR-2+YR-3+HR-3组合脱色速率最快,24 h和48 h脱色率分别为67.08%和97.18%,为最佳复配菌群。相比单一菌株YR-2、YR-3、HR-3的48 h脱色率,该复配菌群脱色率分别提高2.84%、32.63%和63.82%,表明该复配组合微生物(Aneurinibacillus sp.、Stenotrophomonasacidaminiphila、Klebsiella sp.)在染料脱色方面具有协同作用。
2.4 复配菌群的耐受性研究
高碱度和高盐度是微生物高效处理染料废水两大主要限制因素〔5〕。为探究最佳复配菌群对染料废水的普适性,进一步考察了复配菌群对pH及盐度的适应性。
2.4.1 初始pH对复配菌群脱色率的影响
由图 3(a)可知,不同初始pH下,处理组脱色效率显著高于对照组(未添加复配菌群)。初始pH由7.0增加至10.0时,复配菌群对活性艳红X-3B脱色率无显著差异,48 h脱色率均高达94%以上。当pH在7.0~9.0时,对照组具有一定的脱色效率,这主要是由于模拟染料废水未经过灭菌,水中存在其他微生物,在厌氧条件下引起脱色作用;当pH升至10.0时,对照组的脱色效率显著降低至5%以下,高碱性环境显著抑制了其他微生物的生长,而复配菌群对中性及碱性环境具有广泛适应性。进一步跟踪测定对照组及处理组脱色完成后反应液的终点pH,结果如图 3(b)所示。初始pH在7.0~9.0时,对照组对溶液pH有一定的调节能力,最终pH控制在5.7~5.9之间;当初始pH升高至10.0时,对照组溶液最终pH为8.7,调控能力显著降低;而在初始pH为7.0~10.0时,处理组溶液最终pH均控制在6.7~7.0之间,由此可见,处理组能有效调控高碱性环境以利于微生物进行脱色反应。研究表明偶氮染料在降解过程中形成的中间代谢产物(如芳香胺)比原始偶氮染料的碱性更强,会导致染料废水pH升高〔30-31〕。上述复配菌群在碱性环境中仍具有较高脱色率(94%以上)且出水pH较初始pH均有所降低,表明在Aneuriniba-cillus sp.、Stenotrophomonas acidaminiphila和Klebsiella sp.的复配组合中,存在一种或者多种碱性环境下的产酸微生物,其可以有效调控体系pH以适应自身及其他微生物的生长进而协同脱色,并对厌氧脱色过程中可能产生的芳香胺等中间产物具有一定的降解能力。
图3
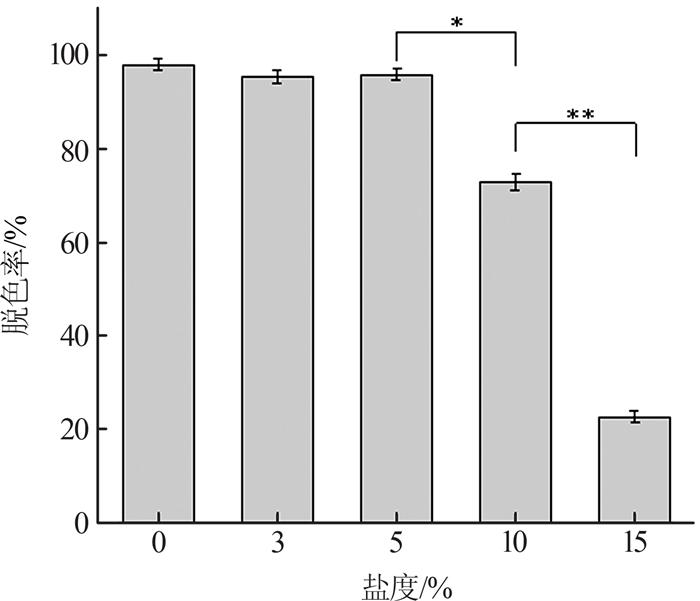
2.4.2 盐度对复配菌群脱色率的影响
图4
由图 4可知,当盐度为0%~5%时,随着盐度的升高复配菌群脱色率并无显著差异,均达90%以上;当盐度为5%~15%时,复配菌群脱色率随着盐度的增加显著降低(p < 0.05),10%盐度时复配菌群脱色率为72.90%,15%盐度时脱色率降低至26.39%。综上,最佳复配菌群在10%盐度下仍可保持70%以上的活力,其耐受盐度范围为0%~10%,属于耐盐菌群。关于耐高盐脱色菌株及菌群的报道较多,大多来源于工业印染废水、制革废水、含盐土壤、海水、海洋沉积物等高盐环境,相同条件下耐盐菌群脱色效率往往比单一耐盐菌株脱色效率更高。A. Khalid等〔37-38〕利用从活性污泥中获得的希瓦氏菌AS96和JM6进行染料脱色研究,结果表明,两个菌株都能够在盐度0.5%~6%条件下对活性黑5、直接红81、酸性红88和分散橙3进行高效脱色,但是随着盐度增加,脱色速率显著降低,当盐度高于6%时,菌株AS96几乎完全丧失脱色能力。谢学辉等〔39〕从印染废水厌氧反应器活性污泥中筛选驯化获得活性黑5的脱色菌群FF,研究表明该菌群具有一定耐盐性,在盐度1%~4%条件下对活性黑5脱色率高达91.0%以上,当盐度为10%时,脱色率降低至38.3%。由此可见,在10%盐度条件下本研究筛选的复配菌群对染料仍具有一定的脱色能力,具有较好的耐盐性能。
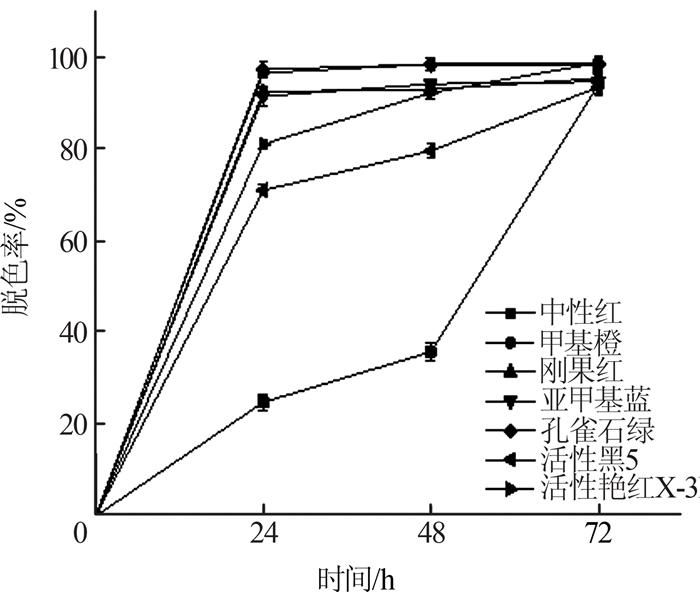
2.5 复配菌群脱色的广谱性评估
图5
由图 5可知,复配菌群对上述不同染料的脱色率均能达到92%以上,具有较好的脱色效果,但脱色速率有所不同。其中,刚果红、甲基橙、亚甲基蓝和孔雀石绿脱色速率较快,24 h脱色率分别为92.55%、96.68%、91.56%和97.44%;中性红、活性艳红X-3B、活性黑5脱色率较慢,脱色率达90%以上分别需要72、48、72 h。研究表明,分子质量低、结构简单、偶氮键数量少、含有促进脱色基团(如—NH2、—OH)的染料更容易被降解脱色,而分子质量高、结构复杂、偶氮键数量多、含有抑制脱色基团(如—SO3-、—OCH3、—CH3)的染料则不容易被降解脱色〔42〕。本研究结果与之相一致,甲基橙、孔雀石绿分子质量低、结构较简单,脱色速率快;刚果红虽然分子质量较高,但是具有更多的促进脱色基团—NH2,有助于其快速脱色;活性艳红X-3B和活性黑5分子质量较高、结构较复杂,脱色速率较慢。中性红和亚甲基蓝都含有杂环结构,但是中性红脱色速率慢,而亚甲基蓝脱色速率快,这可能是由于染料降解还受到氢键、静电作用、范德华力和疏水相互作用等许多因素影响〔43〕。综上所述,复配菌群对不同结构的偶氮染料及不同类型染料均有较好的脱色效果,对于处理高浓度的混合染料废水具有较好的应用前景。
3 结论
(1) 从垃圾渗滤液生化处理系统活性污泥中筛选获得4株高效脱色菌株,分别鉴定为解硫胺素芽孢杆菌Aneurinibacillus sp.YR-2、微嗜酸寡养单胞菌Stenotrophomonas acidaminiphilaYR-3、弗氏柠檬酸杆菌Citrobacter freundii HR-1和克雷伯氏菌Klebsiella sp.HR-3。
(2) 与单一菌株脱色效率相比,复配菌群脱色速度和脱色效率显著提升,最佳复配菌群为YR-2、YR-3、HR-3复配组合(体积比1∶1∶1)。
(3) 复配菌群具有耐碱耐盐特性,pH耐受范围为7~10,盐度耐受范围为0%~10%。
(4) 复配菌群对染料的脱色具有广谱性,对多种偶氮类染料(酸性、直接、碱性、活性)以及杂环类染料和三苯甲烷染料均具有良好的脱色性能,72 h脱色率均达92%以上。
参考文献
Dye removal from water and wastewater using various physical, chemical, and biological processes
[J].DOI:10.5740/jaoacint.18-0051 [本文引用: 2]
Integrated air cathode microbial fuel cell-aerobic bioreactor set-up for enhanced bioelectrodegradation of azo dye Acid Blue 29
[J].DOI:10.1016/j.scitotenv.2020.143752 [本文引用: 1]
Textile finishing dyes and their impact on aquatic environs
[J].
Azo dye decolorization by a halotolerant consortium under microaerophilic conditions
[J].DOI:10.1016/j.chemosphere.2019.125510 [本文引用: 1]
Degradation of azo dye methyl red by alkaliphilic, halotolerant Nesterenkonia lacusekhoensis EMLA3:Application in alkaline and salt-rich dyeing effluent treatment
[J].DOI:10.1007/s00792-017-0918-2 [本文引用: 4]
Removal of azo and anthraquinone dye by plant biomass as adsorbent-A review
[J].
New application of eco-friendly biosorbent giant reed for removal of reactive dyes from water followed by sustainable path for recycling the dyes-loaded sludge in concrete mixes
[J].DOI:10.1007/s10163-020-00998-4 [本文引用: 1]
Biodegradation of some dyes by the green alga Chlorella vulgaris and the cyanobacterium Aphanocapsa elachista
[J].
Optimization of conditions for decolorization of azo-based textile dyes by multiple fungal species
[J].DOI:10.1016/j.jbiotec.2017.08.022 [本文引用: 1]
Degradation and detoxification of azo dyes by a salt-tolerant yeast Cyberlindnera samutprakarnensis S4 under high-salt conditions
[J].
Biodegradation of environmentally hazardous azo dyes and aromatic amines using Klebsiella pneumoniae
[J].DOI:10.1061/(ASCE)EE.1943-7870.0001353 [本文引用: 3]
Decolorization of textile reactive dyes by bacterial monoculture and consortium screened from textile dyeing effluent
[J].DOI:10.1016/j.jgeb.2018.02.005 [本文引用: 1]
Klebsiella and Enterobacter isolated from mangrove wetland soils in Thailand and their application in biological decolorization of textile reactive dyes
[J].DOI:10.3390/ijerph17207531 [本文引用: 1]
Review on electrochemical system for landfill leachate treatment: Performance, mechanism, application, shortcoming, and improvement scheme
[J].DOI:10.1016/j.scitotenv.2020.140768 [本文引用: 1]
Biodegradation of triphenylmethane dye malachite green by Sphingomonas paucimobilis
[J].DOI:10.1007/s11274-008-9941-x [本文引用: 1]
革兰氏染色法观察与区分细菌
[J].DOI:10.3969/j.issn.1004-7549.2019.07.035 [本文引用: 1]
MEGA7:Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets
[J].DOI:10.1093/molbev/msw054 [本文引用: 1]
The neighbor-joining method-A new method for reconstructing phylogenetic trees
[J].
Kinetic characteristics of bacterial azo-dye decolorization by Pseudomonas luteola
[J].DOI:10.1016/S0043-1354(00)00581-9 [本文引用: 1]
Optimization of factors affecting decolorization of sulphonated azo dye Red HE7B in vitro by Bacillus sp. Azo1
[J].DOI:10.18520/cs/v115/i3/505-509 [本文引用: 1]
Decolorization of azo dyes(Direct Blue 151 and Direct Red 31) by moderately alkaliphilic bacterial consortium
[J].DOI:10.1016/j.bjm.2015.11.013 [本文引用: 1]
Klebsiella sp. as nosocomial pathogens: Epidemiology, taxonomy, typing methods, and pathogenicity factors
[J].DOI:10.1128/CMR.11.4.589 [本文引用: 1]
活性黑染料脱色菌的分离鉴定及复合菌群培养研究
[J].DOI:10.3969/j.issn.1000-2375.2019.01.006 [本文引用: 1]
优势菌处理印染废水中水解池的脱色机理
[J].DOI:10.3321/j.issn:1000-6923.1998.05.011 [本文引用: 1]
Biodegradation and detoxification of Direct black G textile dye by a newly isolated thermophilic microflora
[J].
Microbial decolorization and degradation of synthetic dyes: A review
[J].DOI:10.1007/s11157-012-9287-6 [本文引用: 1]
Decolourization of azo dye methyl MTCC red by Saccharomyces cerevisiae MTCC-463
[J].DOI:10.1016/j.chemosphere.2006.12.087 [本文引用: 1]
Biological decolorization of reactive azo dye by anaerobic/aerobic-sequencing batch reactor system
[J].DOI:10.30955/gnj.001493 [本文引用: 1]
Bacterial decolorization and degradation of azo dyes: A review
[J].DOI:10.1016/j.jtice.2010.06.006 [本文引用: 1]
Microbial degradation of dyes: An overview
[J].DOI:10.1016/j.biortech.2020.123728 [本文引用: 1]
Culture of previously uncultured members of the human gut microbiota by culturomics
[J].
Azo dye decolorization by halophilic and halotolerant microorganisms
[J].DOI:10.1007/s13213-010-0144-y [本文引用: 1]
Halophilic & halotolerant prokaryotes in humans
[J].DOI:10.2217/fmb-2017-0237 [本文引用: 1]
Accelerated decolorization of structurally different azo dyes by newly isolated bacterial strains
[J].DOI:10.1007/s00253-007-1302-4 [本文引用: 1]
Decolorization of azo dyes by Shewanella sp. under saline conditions
[J].DOI:10.1007/s00253-008-1498-y [本文引用: 1]
高效混合菌群FF的筛选及其对活性黑5的脱色作用
[J].DOI:10.3969/j.issn.1671-0444.2013.06.019 [本文引用: 1]
Isolation and screening of bacterial isolates from wastewater treatment plants to decolorize azo dyes
[J].DOI:10.1016/j.jbiosc.2017.11.008 [本文引用: 1]
Isolation, identification and application of novel bacterial consortium TJ-1 for the decolorization of structurally different azo dyes
[J].DOI:10.1016/j.biortech.2007.12.074 [本文引用: 1]






 津公网安备 12010602120337号
津公网安备 12010602120337号